近日,南京农业大学沈其荣院士团队在微生物生态学国际权威期刊《The ISME Journal》上发表了题为“Protist predation promotes antimicrobial resistance spread through antagonistic microbiome interactions” 的研究论文,探讨了土壤微生物互作对抗生素抗性动态过程的影响, 揭示了土壤原生动物捕食与抗生素抗性传播之间的复杂关系。

抗生素抗性(Antibiotic Resistance, AR)对全球公共健康造成了严重威胁。长期以来,抗生素的滥用被认为是抗生素耐药基因(Antibiotic Resistance Genes, ARGs)传播的主要驱动因素。然而,越来越多的证据表明,土壤复杂的微生物互作关系可能同样对ARGs的迁移和扩散起着关键作用。
该研究通过多组学分析与室内培养实验相结合,首先对田间土壤样本进行了宏基因组测序分析,发现了土壤原生动物与ARGs直接的线性关系。随后利用宏基因组数据获得了土壤高质量的细菌基因组,并在基因组中识别了潜在的抗生素抗性基因(ARGs)和生物合成基因簇(Biosynthetic Gene Clusters, BGCs),通过这一分析对细菌基因组功能进行了预测,并发现了土壤原生动物丰度与抗生素生产细菌之间呈现显著正相关。此外,室内共培养实验也验证了这一机制,在原生动物捕食压力下,土壤微生物群落中的抗生素生物合成相关基因以及ARGs的表达均显著增加。综合研究结果表明,土壤原生动物捕食使能产生拮抗性次级代谢物的细菌具有竞争优势,从而间接促进了抗生素耐药菌的繁殖。

图1 土壤细菌高质量基因组获取和潜在功能预测
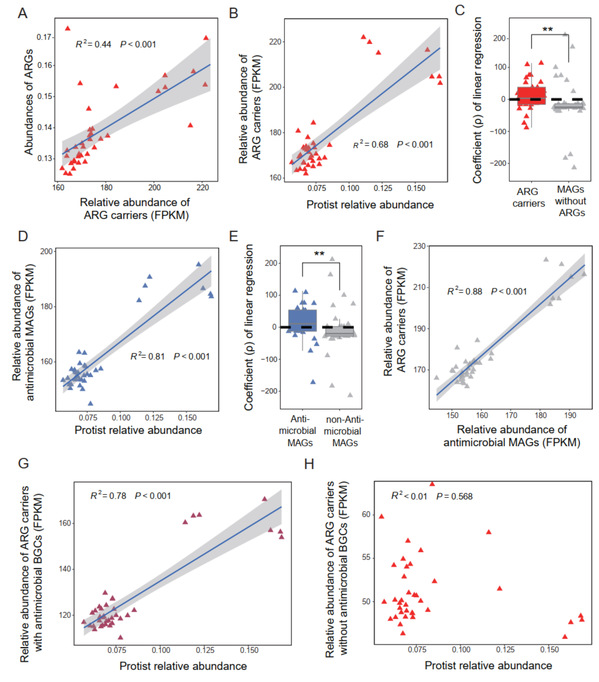
图2 土壤不同功能细菌的相对丰度与ARGs以及原生动物之间的关系
该项研究揭示了土壤生态系统中原生动物捕食行为与抗生素耐药基因扩散之间的潜在关联,不仅丰富了对土壤生态系统中微生物相互作用的理解,也为控制抗生素抗性传播提供了新的理论依据,管理土壤原生动物或许能够成为控制抗生素抗性传播的重要手段。未来的研究将进一步探索如何通过精准调控土壤微生物生态过程(比如土壤原生动物与细菌互作关系)来有效管理抗生素抗性扩散。
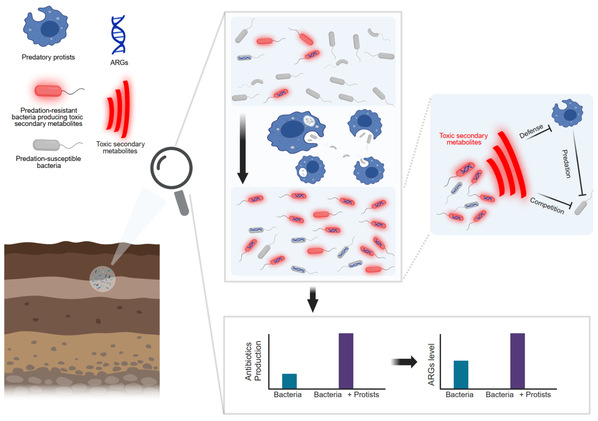
图3 土壤原生动物捕食促进抗生素抗性传播的潜在机制概念图
南京农业大学资环学院刘宸博士生为论文的第一作者,熊武教授为论文的通讯作者,沈其荣院士、韦中教授、王世梅教授、Alexandre Jousset教授、荷兰乌特勒支大学George A Kowalchuk教授和Mohammadhossein Ravanbakhsh博士参与并指导了该研究工作,课题组硕士生王艺锦和周泽源也参与了该研究。该研究得到了国家自然科学基金重大项目等科研项目的支持。
文章信息:
Chen Liu, Yijin Wang, Zeyuan Zhou, Shimei Wang, Zhong Wei, Mohammadhossein Ravanbakhsh, Qirong Shen, Wu Xiong, George A Kowalchuk, Alexandre Jousset, 2024. Protist predation promotes antimicrobial resistance spread through antagonistic microbiome interactions. The ISME Journal, wrae169. https://doi.org/10.1093/ismejo/wrae169
Gaofei Jiang, Chen Liu, Wu Xiong, Qirong Shen, Zhong Wei, 2024. Protist predation selects for the soil resistome. The ISME Journal, wrad007. https://doi.org/10.1093/ismejo/wrad007
