8月24日,南京农业大学生命科学学院环境微生物学团队在《Nucleic Acids Research》上在线发表了题为”DipR, a GntR/FadR-family transcriptional repressor:regulatory mechanism and widespread distribution of thedipcluster for dipicolinic acid catabolism in bacteria”的研究论文,揭示了新型的GntR/FadR家族转录调控蛋白DipR在细菌降解DPA过程中的调控机制。
吡啶-2,6-二羧酸(dipicolinic acid,DPA)是芽孢杆菌等内生孢子抵抗逆境(如热、辐射)的关键组分,占孢子干重的10-15%。由于芽孢在土壤、水体和动植物表面等环境广泛分布,DPA在环境中也无处不在。据测算仅海洋一公里深的底泥中DPA的含量4.6~35×109吨碳元素,占地球生物质碳库的0.8~6%。DPA在孢子萌发后会迅速释放到环境中去。有意思的是,芽孢杆菌不能回收再利用DPA作为营养源。释放的DPA能被其他多种细菌如无色杆菌、产碱杆菌和贪铜菌等降解利用,但其代谢机制仍是未解之谜。近年来,南京农业大学生命科学学院环境微生物学团队发现产碱杆菌Alcaligenes faecalisJQ135可以高效降解DPA,通过18O2稳定同位素标记法测定了DPA的代谢途径,通过转座子随机突变法成功克隆出DPA降解基因簇,命名为“dip”(Int. Biodeterior. Biodegrad. 2021和Appl.Environ. Microbiol. 2022)。然而,细菌降解DPA的转录调控机制尚不清楚。
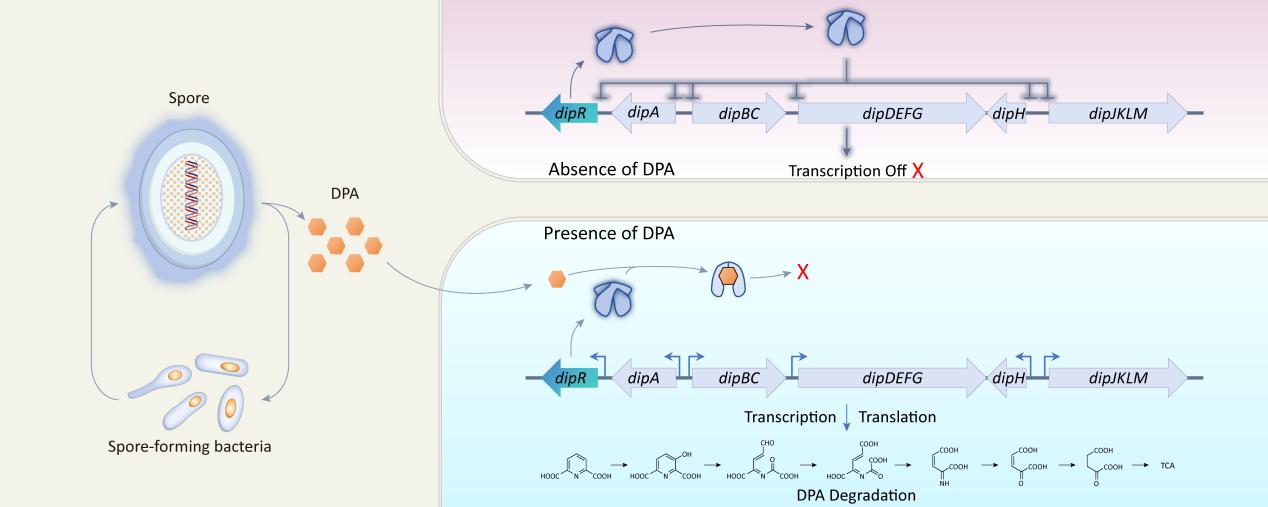
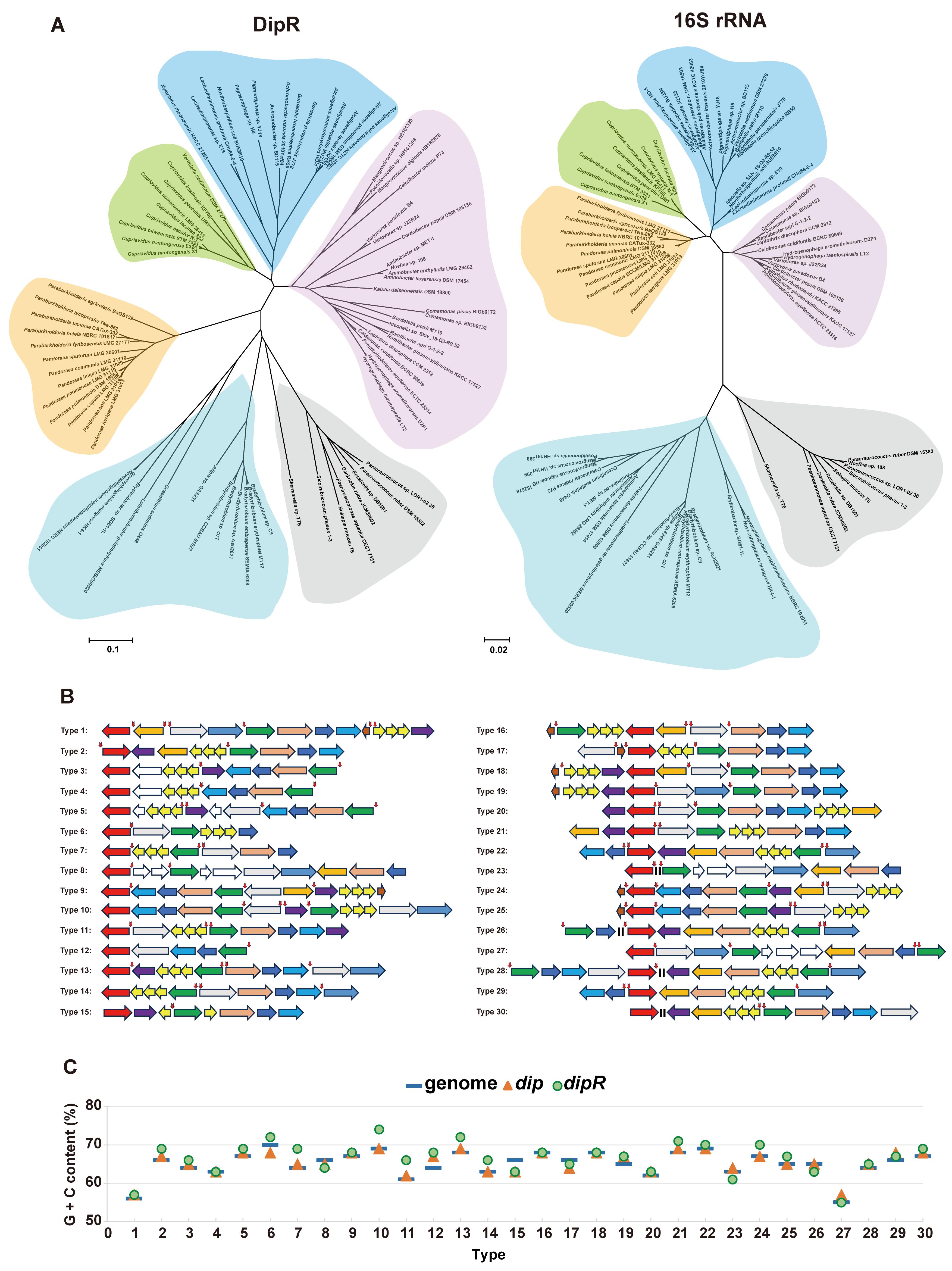
本研究以产碱杆菌Alcaligenes faecalisJQ135为材料,利用RT-PCR、5’-RACE、EMSA、DNaseI Footprinting和氨基酸定点突变等方法系统鉴定了DipR的功能,发现dip簇由dipR、dipA、dipBC、dipDEFG、dipH和dipJKLM等6个转录单元组成;DipR蛋白有6个结合位点共享6 bp保守序列5’-GWATAC-3’,且该序列是DipR结合所必需的;氨基酸残基R63、R67、H196和H218在DipR调控中发挥了重要作用;生物信息学分析表明dip簇广泛存在于39个属、80个种细菌中,并随宿主共进化。本研究揭示DPA降解及转录调控机制,为深入理解环境中微生物降解DPA的分子机制以及基于DPA的地球生物炭库物质循环提供新的理论基础。
南京农业大学生命科学学院博士研究生姜寅虎为论文第一作者,邱吉国副教授为通讯作者,蒋建东教授、何健教授和洪青教授等参与该研究。该研究得到了南京农业大学中央高校基本科研业务费专项资金项目和国家自然科学基金面上项目的资助。
