2月20日,Molecular Plant在线发表了南京农业大学窦道龙团队题为“RLKdb: A comprehensively curated database of plant receptor-like kinase families”的论文。该团队此前开发了基于拓扑结构鉴定各界生物膜受体激酶的注释流程(Yin et al., 2023),该研究进一步开发了标准化的植物RLK生物信息学分析流程,利用该流程在300个具有染色体组装水平的植物基因组上鉴定了~220,000个RLKs,并构建了基于WEB的植物RLK数据库平台(https://biotec.njau.edu.cn/rlkdb)。该数据库为植物RLK提供了全面注释信息,包括家族分类、遗传图谱、基因模型、蛋白功能结构域、转录因子结合位点、PCR引物设计、3D结构、互作蛋白、以及物种分布等。此外,数据库还提供了在线鉴定和其他个性化分析工具。总之,该数据库为植物RLK功能研究和全景式进化分析研究提供了一站式数据支持。
植物类受体激酶(RLKs)是一类重要的植物膜蛋白,包含有胞外结构域、跨膜结构域和胞内激酶结构域。自从第一个RLK基因ZmPK1从玉米中被克隆出来以来(Walker and Zhang, 1990),研究人员已经在多种植物中鉴定和研究了许多RLK基因。这些研究表明,RLKs在植物的生长发育、激素信号传导以及抗病和抗逆境等方面都发挥着重要的作用(Dievart et al., 2020)。
鉴于RLKs共享保守的单系起源的激酶结构域,根据其胞外结构域的不同可划分为十几个家族(Shiu & Bleecker, 2001)。以往,基于序列同源性的方法被广泛用于RLK家族的鉴定和分类,但此分类方法会漏掉具有类似结构的远缘序列和潜在的新家族。因此,业界不仅亟需一个准确可靠RLK鉴定、分类工具,而且还需要一个专业数据库为重要植物RLK基因研究提供支撑。

图1. RLKdb主页
RLKdb有三个访问接口:
1)RLKs:通过植物基因组对应的RLKome和RLK家族分类,浏览植物RLK基因;
2)Tools:通过序列和结构检索访问RLK,并提供在线注释RLKome功能;
3)Stats & Download:提供RLK家族统计和对应序列和注释信息下载。
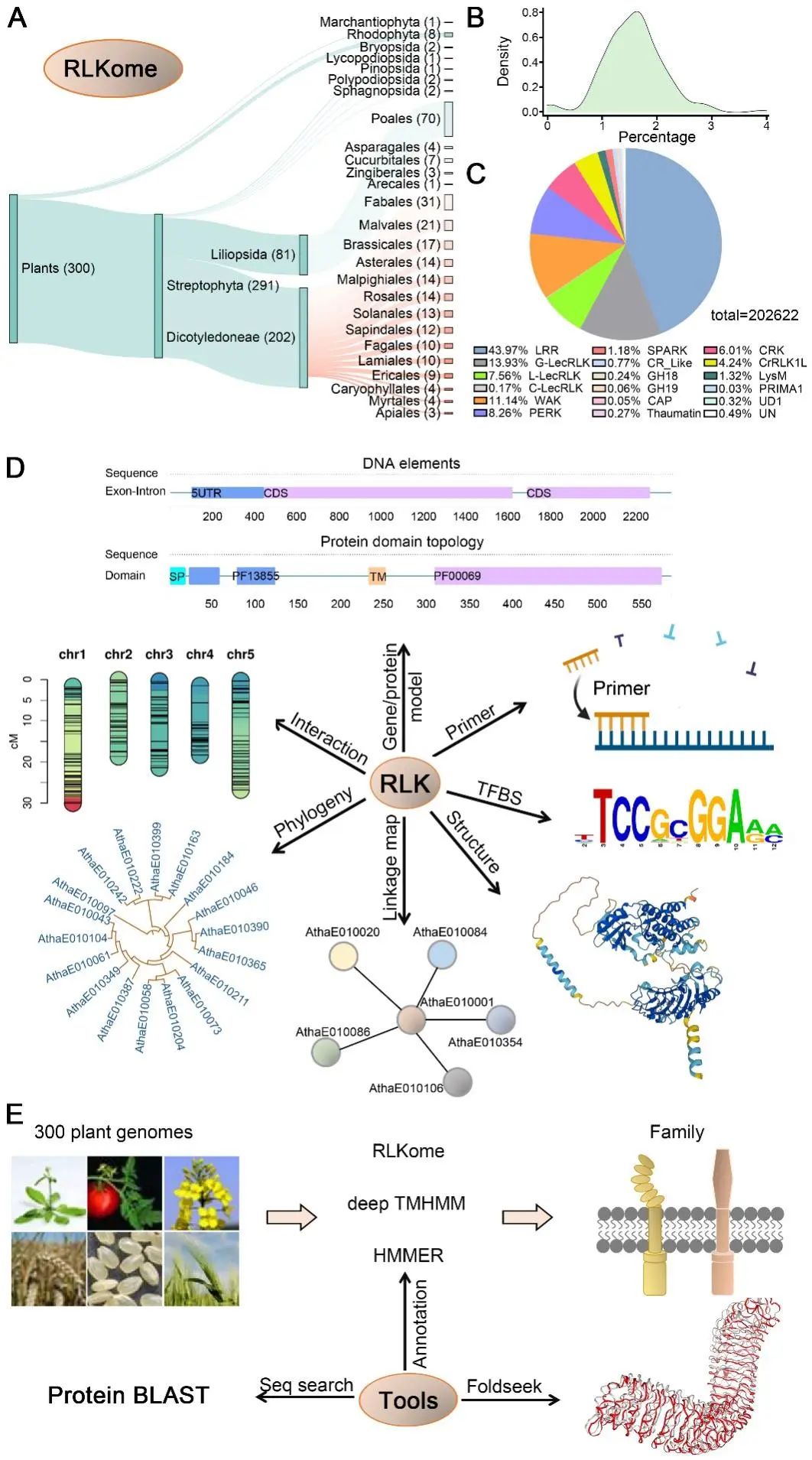
图2. RLKdb主要模块及功能
南京农业大学植物保护学院尹志远副教授为论文第一作者,前沿交叉研究院刘金定副教授为论文通讯作者,团队负责人窦道龙教授参与并在数据库构建中给予重要指导。本研究得到国家自然科学基金、国家中药材产业技术体系和江苏省自然科学基金等项目的支持。

