7月21日,生物信息领域重要期刊Briefings in Bioinformatics在线发表了南京农业大学人工智能学院计智伟教授课题组的题为“HNSPPI: A Hybrid Computational Model Combing Network and Sequence Information for Predicting Protein-Protein Interaction”的研究论文。在这项工作中,研究人员借鉴了自然语言处理技术,开发了一种轻量级的蛋白质语言模型HNSPPI,用于预测蛋白-蛋白相互作用。

HNSPPI通过整合氨基酸序列信息和PPI网络的拓扑特性,综合表征任一蛋白对之间的内在关系(图1)。研究表明,HNSPPI在人类、酿酒酵母、小鼠等六个基准数据集上表现卓越,预测性能明显优于其他五种现有算法(包括DeepFE-PPI、DeepPurpose等)。最后,HNSPPI模型被用于探索SARS-CoV-2-Human相互作用系统,推理出3个与病毒蛋白S或M有潜在互作关系的宿主蛋白。总之,HNSPPI是一种很有前途的AI模型,可用于鉴定病毒/病原菌入侵宿主后的靶向结合蛋白。

图1. HNSPPI模型计算流程图。
研究人员首先开展消融实验,证明了特征融合策略的重要性。他们测试了HNSPPI集成模型、仅用Net2vec模块、仅用Seq2vec模块在六个公共数据集上的预测性能。从图2可见,HNSPPI集成模型在人类、酿酒酵母、黑腹果蝇、幽门螺杆菌等四个PPI数据集上的性能显著优于仅使用Net2vec模块或Seq2vec模块的预测模型。
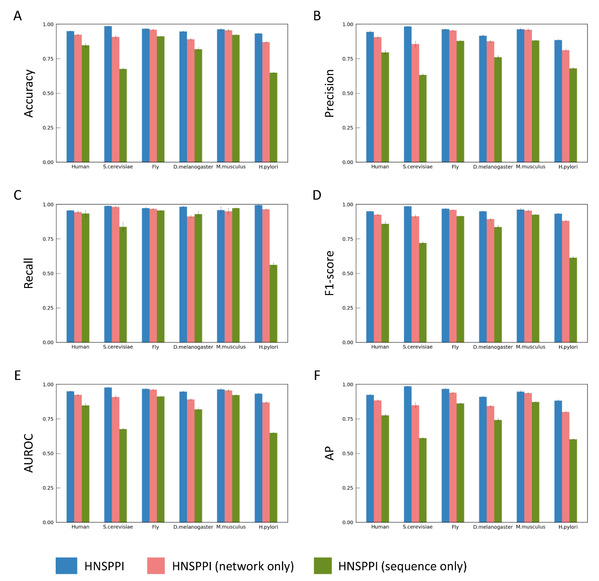
图2. HNSPPI模型在六个公共数据集上的消融实验
进一步,研究人员在所有六个基准数据集上将HNSPPI模型与其他5种现有算法进行了性能比较。总体而言,HNSPPI 在所有测试数据集上提供了最佳性能。特别地,HNSPPI在酿酒酵母(图3)、黑腹果蝇、幽门螺杆菌等数据集上的表现显著优于其他算法。
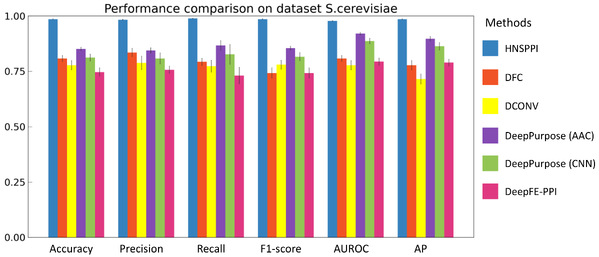
图3. 所有算法在酿酒酵母PPI数据集上的性能比较
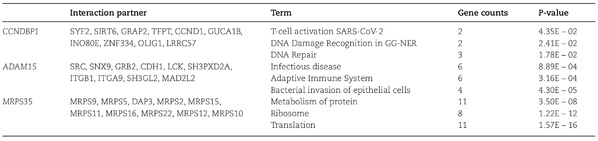
为了展示该项研究的应用前景和可扩展性,研究者将HNSPPI模型应用于SARS-CoV-2-Human相互作用的复杂系统,并预测了与关键病毒蛋白S或M有潜在互作关系的3个宿主蛋白CCNDBP1, ADAM15, MRPS35(图4A-B)。进一步,他们分析了感染Omicron毒株BA.1和BA.2患者的转录组数据,暗示了这些潜在的相互作用可能与T细胞激活和适应性免疫反应相关(表1)。
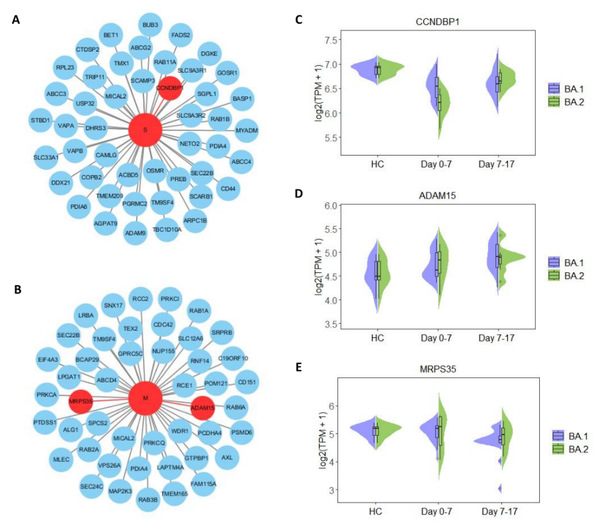
图4. SARS-CoV-2-Human互作系统的案例研究
表1. CCNDBP1、ADAM15和MRPS35相互作用蛋白的功能注释
综上所述,该研究提出了一种用于PPI预测的新型计算框架 HNSPPI。通过融合蛋白质序列和相互作用网络拓扑的特征信息,HNSPPI提供了跨物种的显著预测性能。与当前五种最先进的算法相比,该模型在PPI预测方面实现了卓越的性能提升。与诸多深度学习模型不同的是,HNSPPI提供了轻量级的特征提取策略,并通过简单的分类器即可实现预测。这些优势保证了HNSPPI模型的高可解释性和计算效率。总之,HNSSPPI有望在不久的将来被广泛应用于探索各种病毒-宿主相互作用系统。
本文的第一作者为南京农业大学人工智能学院硕士生谢诗婕,通讯作者为计智伟教授。谢小军博士为算法优化提供了技术支持。首都医科大学赵昕教授、南京农业大学刘斐教授、王一鸣教授、平继辉教授参与了本项研究工作。感谢南京农业大学海外高层次引进人才启动项目、江苏省自然科学基金项目、科技部外专项目、中央高校基本业务经费等项目的支持。
