2022年2月,理学院张良云教授团队在生物信息学领域国际权威期刊《Briefings in Bioinformatics》上在线发表了两篇论文。
论文一标题为“MiRLoc: predicting miRNA subcellular localization by incorporating miRNA–mRNA interactions and mRNA subcellular localization”。该研究构建了一个用于预测miRNA的亚细胞定位的计算框架(MiRLoc),并为生物分子亚细胞定位的预测研究提供了一种重要参考。

众所周知,miRNA 在细胞的不同部位具有特定的生理作用。因此,miRNA的亚细胞定位对于了解其生理功能至关重要。近年来,在经典的原位杂交技术(ISH) 和高通量RNA测序技术的基础上,人们开发了许多检测RNA分子亚细胞定位的方法。然而,这些基于“湿实验”方法既昂贵且耗时,促使了计算方法的快速发展。
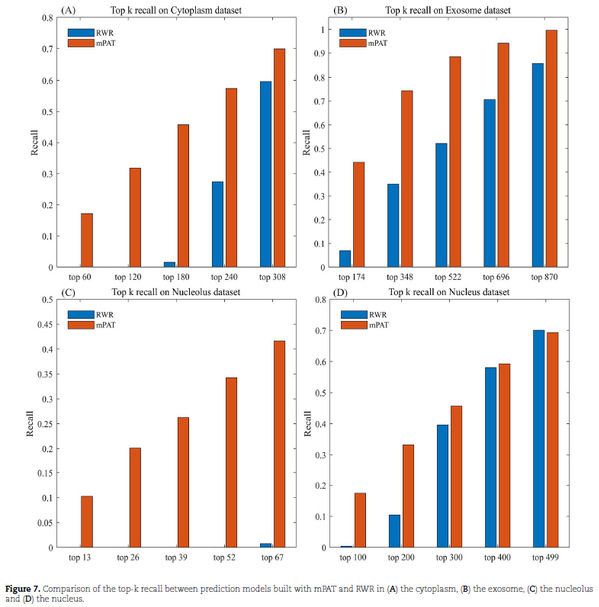
在该研究中,作者基于功能相似的miRNA倾向于定位到相同的亚细胞单位假设,以miRNA功能相似网络为背景,使用随机游走算法(RWR)将已知的miRNA亚细胞定位作为种子信息,通过背景网络将亚细胞定位信息传播给候选miRNA,从而预测miRNA的亚细胞定位。并且在多个亚细胞数据集上获得了较为可观的预测性能。其次,基于miRNA靶标mRNA的亚细胞定位数据有助于预测miRNA的亚细胞定位假设,作者进一步提出了信息传递算法(mPAT) 改进预测模型。结果表明,mPAT算法优于RWR算法,尤其在不平衡数据集上的优越性能更好。
博士研究生许明敏为论文第一作者,骈聪副教授﹑张良云教授为论文通讯作者,陈园园副教授与南京先声诊断有限公司的许志辉博士参加了该文研究。该研究得到国家自然科学基金以及南京农业大学高级人才启动基金的资助。
文章链接:https://doi.org/10.1093/bib/bbac044
论文二题目为“Adapt-Kcr: a novel deep learning framework for accurate prediction of lysine crotonylation sites based on learning embedding features and attention architecture”。该研究构建了一个有效的深度学习模型,用来预测蛋白质赖氨酸巴豆酰化等。
蛋白质赖氨酸巴豆酰化(Kcr)是在真核生物中发现的一种新的蛋白翻译后修饰(PTM),并由巴豆酰转移酶和去巴豆酰化酶共同调节。目前,已知Kcr 参与DNA 复制、精子发育和胚胎干细胞分化,并与急性肾损伤、HIV 潜伏期和结肠癌相关。因此,有效识别Kcr 位点是至关重要的。然而,现有的检测Kcr 位点的实验技术成本高,迫切需要一种新的计算方法来解决这个问题。
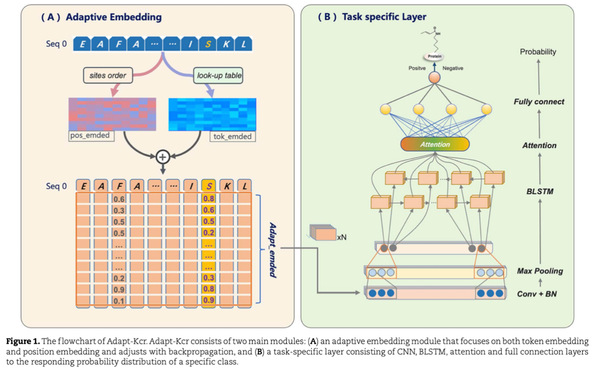
作者首次提出了一种先进的深度学习模型(Adapt-Kcr)用于蛋白Kcr位点的识别。在独立测试集上,Adapt-Kcr 的表现优于目前最先进的Kcr 预测模型,准确度提高了3.2%,受试者工作特征曲线下面积提高了1.9%。与其他Kcr 模型相比,Adapt-Kcr 还具有更强的区分巴豆酰化和其他赖氨酸修饰的能力。此外,作者基于相同架构也可以有效预测SARS-Cov-2中的磷酸化位点。结果表明,本文提出的Adapt框架(包括学习嵌入特征和注意力架构)不仅可以预测蛋白Kcr位点,还具有预测其他蛋白质翻译后修饰位点的强大潜力。

南京农业大学博士研究生李祖潭和博士研究生房婧雅为论文共同一作,南京农业大学骈聪副教授、张良云教授、陈园园副教授为论文通讯作者。该研究得到国家自然科学基金和南京农业大学人才启动基金的资助。
