近日,我校前沿交叉研究院作物表型组学交叉研究中心周济实验室,在国际植物科学顶级期刊《植物生理学》(Plant Physiology,IF = 8.34)中的突破性技术、工具和资源(Breakthrough Technologies, Tools, and Resources)专栏,在线发表了最新的作物表型研究型论文——Large-scale field phenotyping using backpack LiDAR and CropQuant-3D to measure structural variation in wheat。
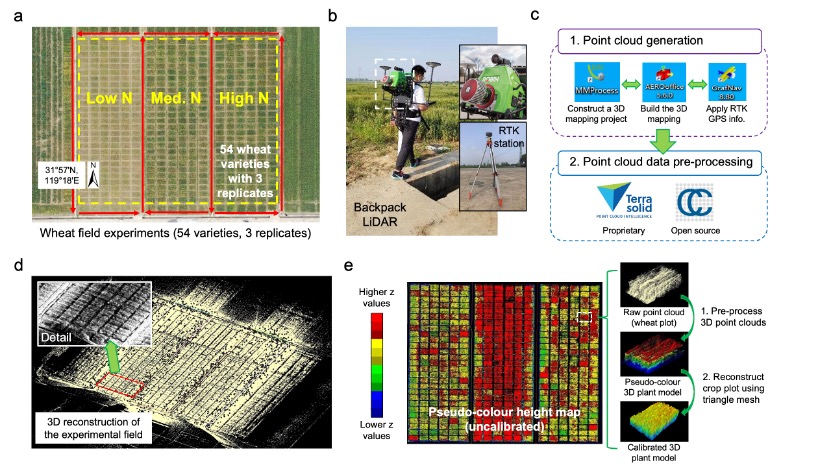
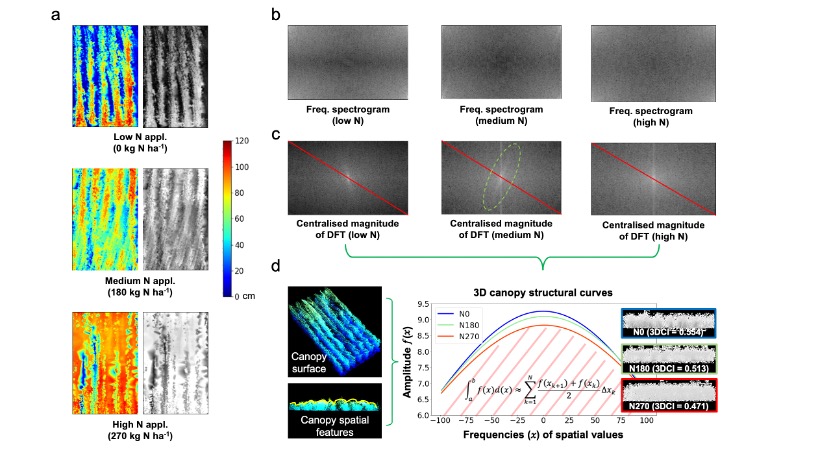
论文结合了背包式激光雷达、三维计算机视觉技术和开源图像分析算法开展了大规模田间小麦表型性状采集,建立了三维表型分析平台CropQuant-3D,进而通过田间获取的氮素响应性状来鉴选不同的氮素高效利用小麦品种。在作物研究中首次使用了背包式激光雷达在大规模田间试验中开展表型采集技术手段,通过开源三维表型分析算法对获取的数以亿计的三维点云进行了自动化分析,量化了不同小麦品种在田间的空间形态特征和相应的关键农艺性状,进而获取了这些性状与产量、氮肥响应之间的联系。本研究通过结合离散傅立叶变换(Discrete Fourier Transform)和三维空间特征转换提出了精确测量不同小区内作物高度、解析复杂冠层结构的算法,通过11个小麦品种结合高中低三个施氮水平在总计近1公顷田间试验中验证了算法的稳定性和鲁棒性,解析了表型性状和小麦氮素响应、产量构成因素和小区产量等关键性状的关系。此外,研究还着重介绍了周济实验室自主开发的开源软件系统CropQuant-3D的性状分析算法及其延展性,通过对无人机、龙门架等不同设备获取的三维点云数据的统一分析,展示了该平台在大规模田间表型分析和开源共享等方面的巨大潜力。
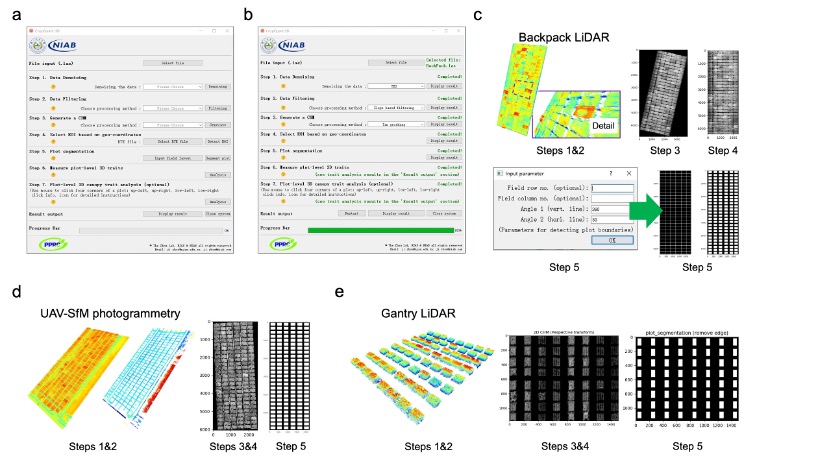
随着测序技术和基因组学的飞速发展,植物遗传学飞速发展,基因型数据海量扩充。大规模可靠表型数据的匮乏已成为解析遗传信息、精准育种及高效栽培管理的一大瓶颈。这对高通量、多环境、多生育期且高可靠性的表型组学研究提出了新的要求。如何结合快速发展的包括激光雷达技术在内的多尺度遥感和智能化数据解析有望为大规模育种、栽培管理和农业生产提供重大的技术支持。本研究使用的背包激光雷达较好满足了以上需求,且兼具易使用、易运输和用户友好等优势,克服了其他表型平台的在多地点、大规模数据采集上的局限。自主开发的CropQuant-3D分析平台也实现了在普通计算机上快速完成对约1公顷的小麦试验的性状提取,为表型性状采集、鉴定这一瓶颈的提供了解决方案,也为作物研究中进一步高效挖掘作物关键基因提供了表型支持。
我校周济教授为本文的通讯作者,周济实验室的朱宇磊硕士(工学院)为第一作者,孙港硕士(农学院)、丁国辉博士生(农学院)和周洁硕士等实验室成员也参与了部分工作。我校作物遗传与种质创新国家重点实验室、前沿交叉研究院以及江苏省现代作物生产协同创新中心同为第一通讯单位。我校丁艳锋教授、金时超副教授也参与了本项目。此外,英国国立农业植物研究所的剑桥作物研究中心(Cambridge Crop Research, National Institute of Agricultural Botany, NIAB)、中国科学院分子植物科学卓越创新中心和江苏丘陵地区镇江农业科学研究所也共同参与了项目研发。
文章链接:https://doi.org/10.1093/plphys/kiab324

